本站讯 日前,中国海洋大学食品科学与工程学院梁兴国教授团队讲师安然和博士生李琳等在核酸领域顶级刊物Nucleic Acid Research(《核酸研究》)上刊登了题为“Nonalternating purine pyrimidine sequences can form stable left-handed DNA duplex by strong topological constraint”(非嘌呤嘧啶交替序列可以在强拓扑限制下形成左手双螺旋DNA)的研究论文。该研究利用自主开发的单链DNA环化以及两条互补的单链DNA杂交形成左手双螺旋的技术,详细研究了普通DNA序列形成左螺旋DNA的情况,并得出了“任何序列都可以形成稳定左螺旋DNA”这一打破传统认知的结论。
早在上世纪70年代,以Alexander Rich为代表的科学家就发现并确认了左螺旋DNA(Z-DNA)的存在,但由于只有d(CG)n这种嘌呤和嘧啶交替(APP)的序列在高盐浓度、化学修饰或较强的负超螺旋等特殊条件下会形成左螺旋DNA,其普遍的生物学意义一直受到质疑。科学家们对于普通的DNA序列是否会形成左螺旋DNA有着各种猜测,但一直没有直接的技术手段进行研究。同时,大家对于在体内APP序列会形成Z-DNA的本质也缺乏足够的认识。近五年来,梁兴国教授团队在DNA和RNA环化以及构建生理条件下稳定左螺旋DNA方面取得了重大突破。本研究就是在这些成果的基础上进行探索,回答了困扰大家多年的“左螺旋DNA形成的本质”这一基础科学问题(图1),也为解释分子进化过程中为什么选择了D-ribose和D-deoxyribose(D构型的核糖和D构型的脱氧核糖)提供了重要线索。
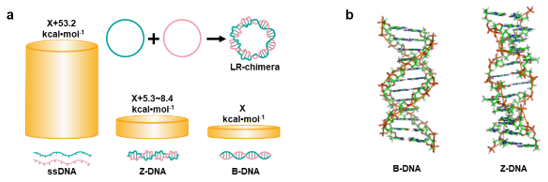
图1.(a)拓扑限制法制备左螺旋DNA示意图及相同序列的线性ssDNA、Z-DNA和B-DNA自由能水平的比较;(b)右螺旋(B-DNA)和左螺旋(Z-DNA)的结构模型
本研究通过特异性抗体结合、熔点测定、酶切以及反映分子手性特征的圆二色性色谱(CD)分析发现,在拓扑限制下普通的序列会形成同APP序列这种特殊序列同样稳定的左螺旋DNA。如图1所示,相对于单链状态而言,互补的DNA序列可以形成自由能大大降低的Z-DNA(左螺旋)和B-DNA(右螺旋)两种状态,而两种状态之间自由能相差很小。在体内APP序列会优先形成左螺旋DNA的原因是,APP序列的左螺旋和右螺旋两种状态的自由能相对于非APP序列更为接近。就像水总倾向于向低处流一样,分子也总是倾向于以自由能更低的状态存在。在没有APP序列的区域,非APP序列就会首当其冲,在适当的拓扑限制下形成相对稳定的左螺旋DNA结构。
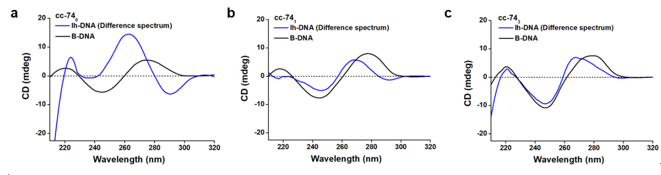
图2.(a)APP序列的左螺旋和右螺旋的圆二色性色谱;(b,c)普通序列的左螺旋和右螺旋的圆二色性色谱
本研究的另外一个重大贡献是发现普通序列左螺旋的CD色谱(表示物质对于各种波长的左旋光和右旋光吸收程度的差异)完全不同于APP序列(图2)。APP序列会在260 nm附近产生较强正值的CD信号(图2a),而普通序列则具有较弱的,同右螺旋DNA更为接近的CD(图2b和c)。这一发现纠正了认为d(CG)n的CD是Z-DNA的特征图谱的错误认识。实际上,同B-DNA的CD一样,Z-DNA的CD也会随序列而产生很大变化。
本研究成果提示,在转录过程中产生的较高强度负超螺旋可能通过形成左螺旋DNA的方式得以暂时缓解,以避免易于造成损伤的较长单链DNA形成。有意思的是,2021年11月在Cell杂志发表的(题为“Z-form extracellular DNA is a structural component of the bacterial biofilm matrix”,DOI: 10.1016/j.cell.2021.10.010)研究表明,细菌会在其表面利用左螺旋Z-DNA形成生物被膜,使细菌产生脱氧核酸酶抗性,为解决或预防生物被膜介导的疾病/感染提供了新思路。结合本研究的发现可以判断,这些胞外Z-DNA应该主要由非APP序列形成,说明左螺旋DNA可能在自然界普遍存在。这让我们再一次认识到,生命会利用一切可能来进行自我保护并提高生存竞争力。在本研究的基础上,我们有望进一步认识左螺旋DNA的形成机制、左螺旋DNA特异性结合蛋白的作用机理以及开发治疗Z-DNA相关疾病的药物等。
该研究由中国海洋大学和青岛海洋科学与技术试点国家实验室合作完成,得到了青岛市高校共建基础发展基金、国家自然科学基金和山东省自然科学基金等项目的资助。
通讯员:李琳 朱尚清
文章链接:https://doi.org/10.1093/nar/gkab1283
团队发表的DNA和RNA环化以及制备稳定左螺旋DNA的论文连接:
1.https://doi.org/10.1093/nar/gkx553
2.https://doi.org/10.1093/nar/gky769
3.https://doi.org/10.1021/jacs.8b13855
4.https://doi.org/10.1093/nar/gkaa181

